Las bacterias verdes del azufre son un grupo bacteriano Gram (-) fotolitoautótrofo metabólicamente especialista, pertenenecen a la clase Chlorobia, Phyllum Bacteroidotas, caracterizadas por ser anaerobias estrictas .
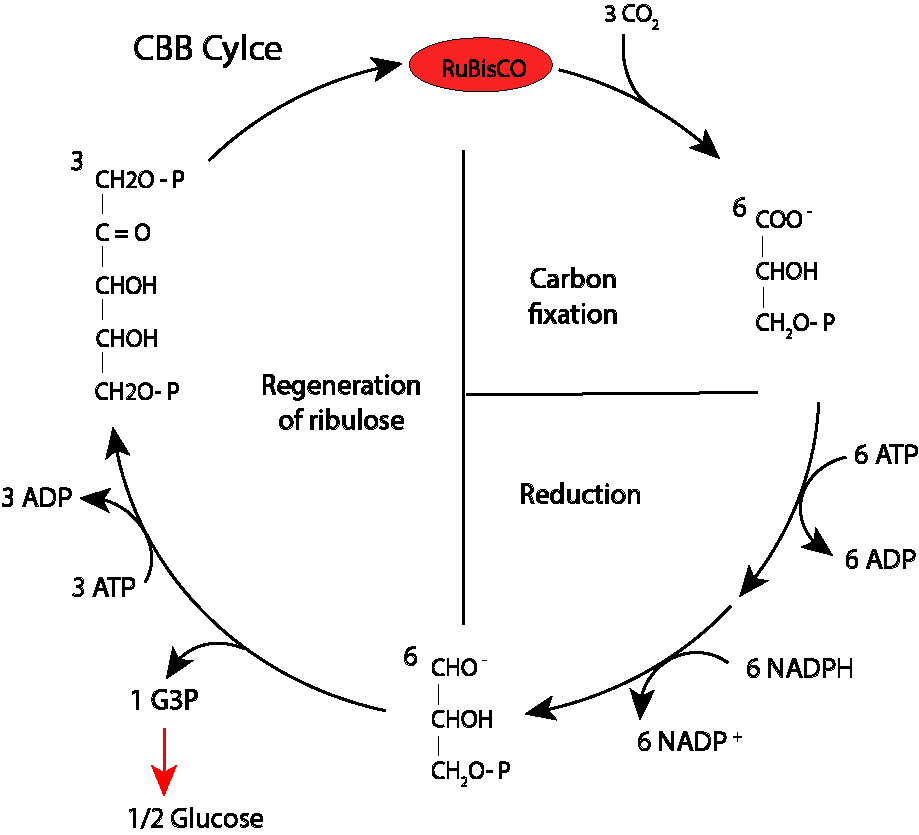
Propias de ambientes anóxicos en fondos de lagos con alto contenido de azufre, utilizan H2S y en menor medida tiosulfatos y So elemental como donores de electrones, carecen del ciclo reductor de Calvin-Benson, en reemplazo fijan CO2 mediante el ciclo reverso del ácido tricarboxílico.
Su aparato fotosintético contiene complejos antena altamente eficientes denominados clorosomas, los cuales les permiten prosperar a bajísimas intensidades de luz, siendo de hecho el grupo fotosintetizador con menores requerimientos de energía lumínica reportado a la fecha.
Este grupo bacteriano utiliza el centro de reacción RC para producir reductantes fuertes, ATP y NADPH, mediante la oxidacion de los compuestos del azufre anteriormente mencionados.
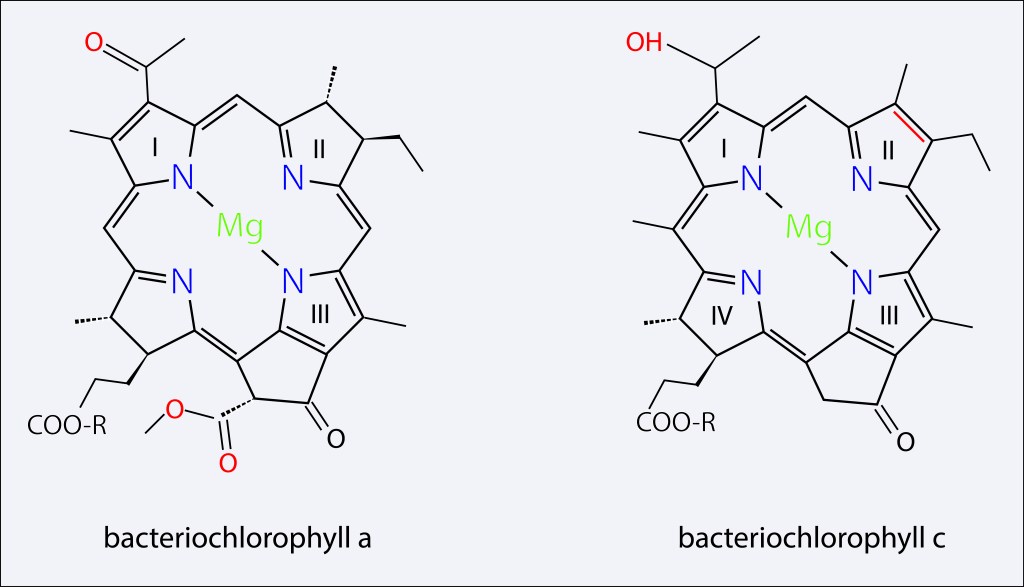
Para activar la cadena de transporte de electrones, RC contiene como cofactor prostético un par de bacterioclorofilas BChla exitable a 840 nm, que les permite pasar de un estado GND un estado exitado P840*, ver Fig. 1.

Figura 1. Potenciales REDOX en la cadena ETC de las bacterias verdes del azufre.
P840 es indispensable para:
- Recepción de energía lumínica proveniente de los complejos antena
- Separación de cargas
- Transferencia de electrones a clusters de proteinas-ferrodoxinas Fe–S, a niveles de energía mucho menores (mas negativos) que los requeridos por las quinonas de bacterias púrpura, lo que permite reducir directamente NAD+.
ETC transfiere la energía desde el par BChla exitado hacia FeSA/B, y finalmente a ferrodoxinas Fd citoplasmáticas altamente reductoras, (514-584 mV) , lo que proporciona potenciales suficientes para reducir NADP + vía ferrodoxina/NADP+ oxidoreductasa, lo que habilita las cadenas memtabólicas responsables de la fijación de carbono.
En cuanto a la síntesis de ATP, a la fecha no hay claridad sobre la ruta responsable de generación de la PMF requerida para la activación de ATP synthase.
En primer lugar, en la cadena de transporte de electrones no participan de manera directa quinonas, y solo menaquinona MK7 está presente en lo que prodría definirse como pool de quinonas.
Por otro lado, a pesar de que GSB contiene genes codificantes de Cyt b y Rieske Fe/S (PetB y PetC respectivamente), no ha sido descrito a la fecha genoma codificador de Cyt c1 y Cyt f , de manera que una ruta cíclica equivalente a la vía Cyt bc1 de bacterias púrpura no ha sido demostrada para GSB. El rol de Cyt b y Rieske Fe/S en el complejo es sujeto de investigación.
De manera alterna, es posible que la acción combinada de ferredoxina como agente reductor de MK7 y NADP deshidrogenasa como fuente de protones, ver Fig.2., permita generar MKOH, de modo que la translocación de protones en una etapa oxidativa subsiguiente de MKOH -> MK7 para síntesis de ATP pueda ocurrir . La reducción de MK7 puede tambien provenir de FeS o la fuente de electrones del sistema. Independiente del agente reductor, no ha sido posible caracterizar complejos oxidantes de MKOH que soporten la teoría, así que la ruta de generación de PMF y retorno de electrones al sistema en alguna vía alterna a Cytc1-Cyt C soluble también está por aclararse. En la Fig 2., las líneas punteadas representan rutas probables no demostradas.
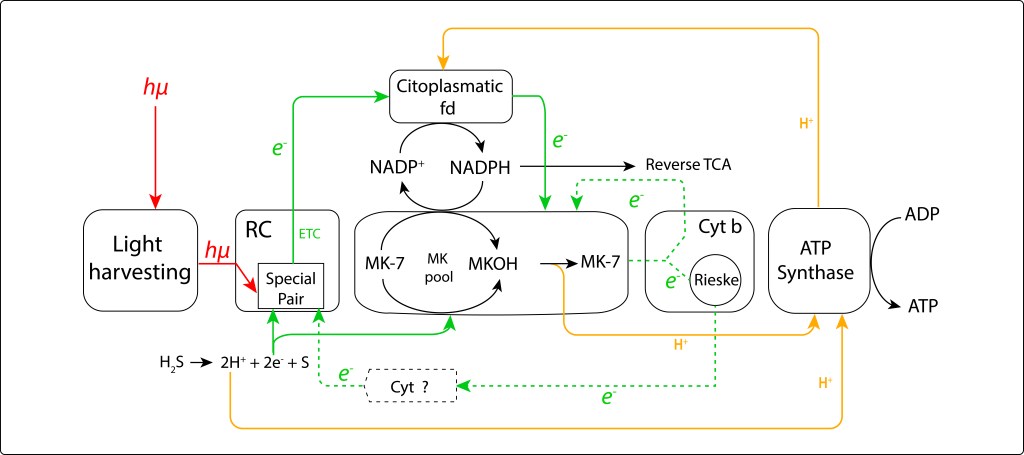
Figura 2. Diagrama de flujo de las probables rutas fotosintéticas en GSB, incluyendo consumo de NADPH para síntesis de ATP. Líneas punteadas indican rutas no demostradas.
Para complementar el tema, la Fig 3 describe la ruta de síntesis de NADPH en bacterias púrpura mediante el consumo de ATP.
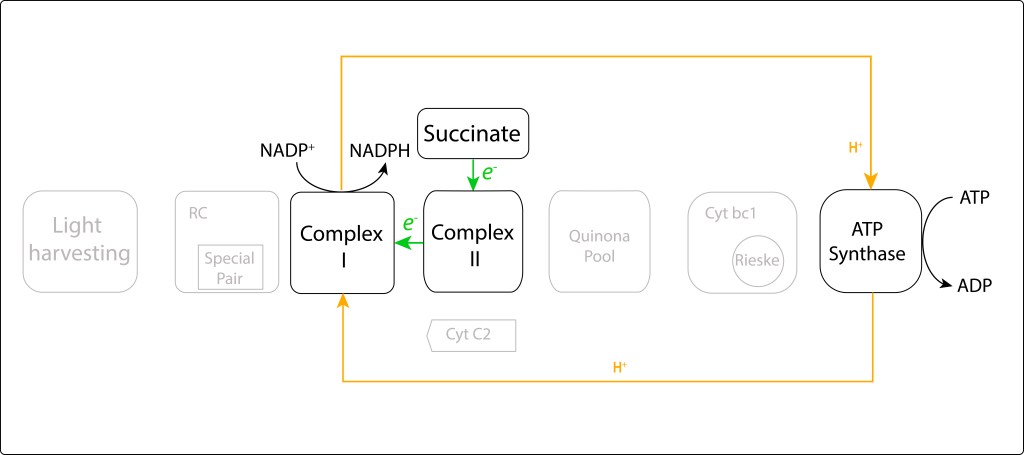
Figura 3. Diagrama de flujo de la síntesis de NADPH a partir de consumo de ATP en bacterias ´púrpura
En resumen, mientras que en bacterias púrpura, debido a su naturaleza débilmente reductora, se hace necesario “invertir” parte del ATP producido para sintetizar NADPH, en GSB, dada su naturaleza fuertemente reductora pero débilmente oxidante, una fracción de NADPH es la que se debe utilizar durante la síntesis de ATP.
Puesto que la incorporación de carbono a partir de CO2 depende de la disponibilidad tanto de NADPH como de ATP en las rutas reductoras (Ciclo de Calvin-Benson y TCA Reverso, entre otros) , y puesto que parte de estos agentes debe ser direccionado hacia la síntesis de los mismos, los aparatos fotosintéticos de Bacterias púrpura y GSB tienen en común una eficiencia limitada. La situación en bacterias púrpura y GSB recuerda la paradoja de la serpiente, quien para sobrevivir, requiere alimentarse de sí misma.
Figura 4. Paradoja de la serpiente
Hace unos 2,000 millones de años, en un proceso de endosimbiosis bioquímica, dos grupos bacterianos, con rutas bioquímicas similares a las mencionadas, fusionaron de alguna manera sus maquinarias fotosintéticas dando origen a un único complejo fuertemente oxidante, fuertemente reductor, lo que por un lado eliminó la necesidad de consumo de productos reductores para síntesis de sí mismos, y por otro lado habilitó la utilización de H2O como una nueva e ilimitada fuente de electrones.
Las consecuencias de dicha convergencia evolutiva marcaron un punto de inflexión en la vida en el planeta, pero esto será tema para las próximas lecturas.
Finalmente, y para cerrar la lectura, la figura 5 desribe de manera resumida el aparato fotosintético en GSB.
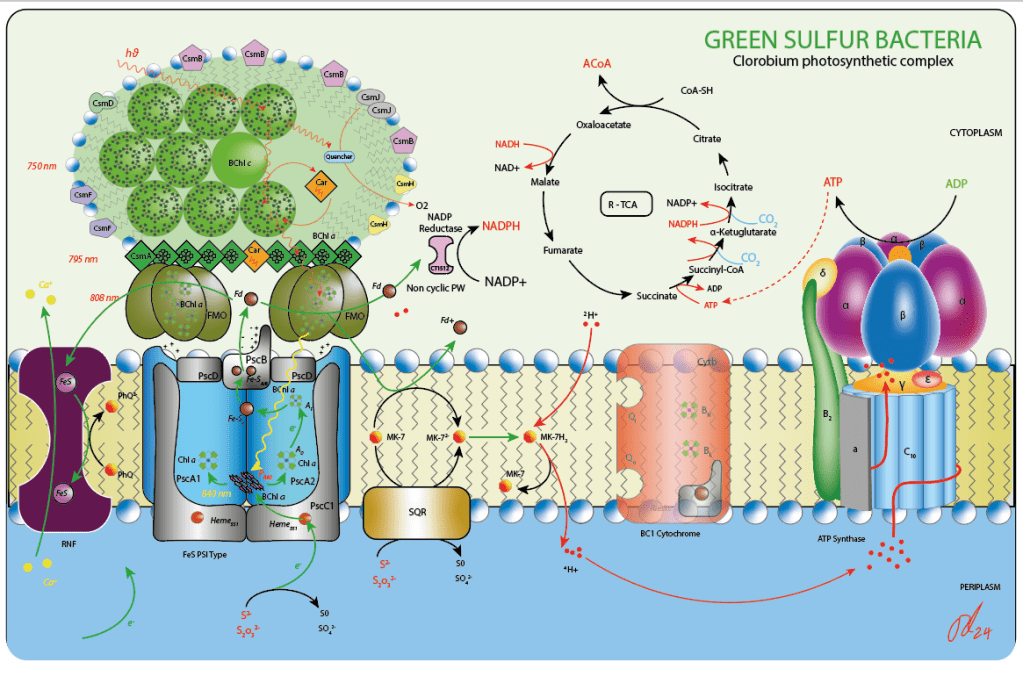
Figura 5. Aparato fotosintético en bacterias verdes del azufre


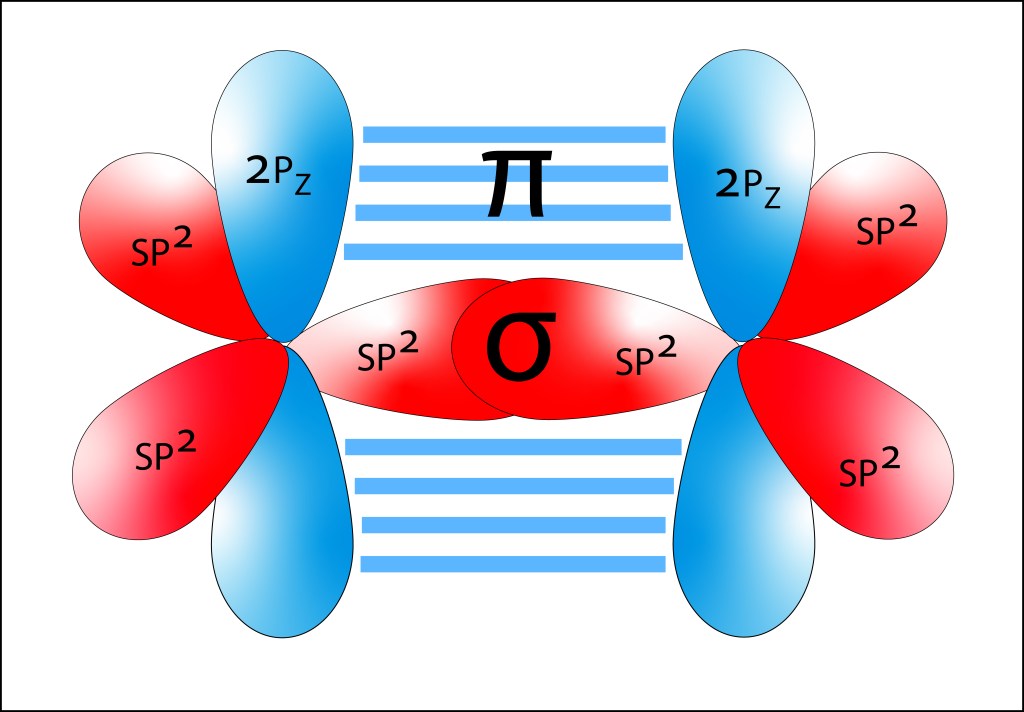
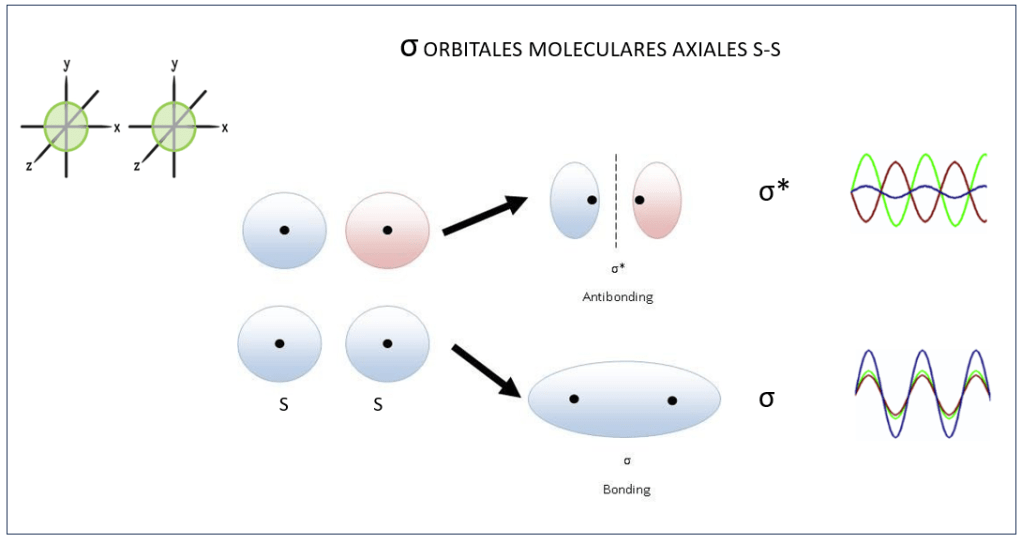
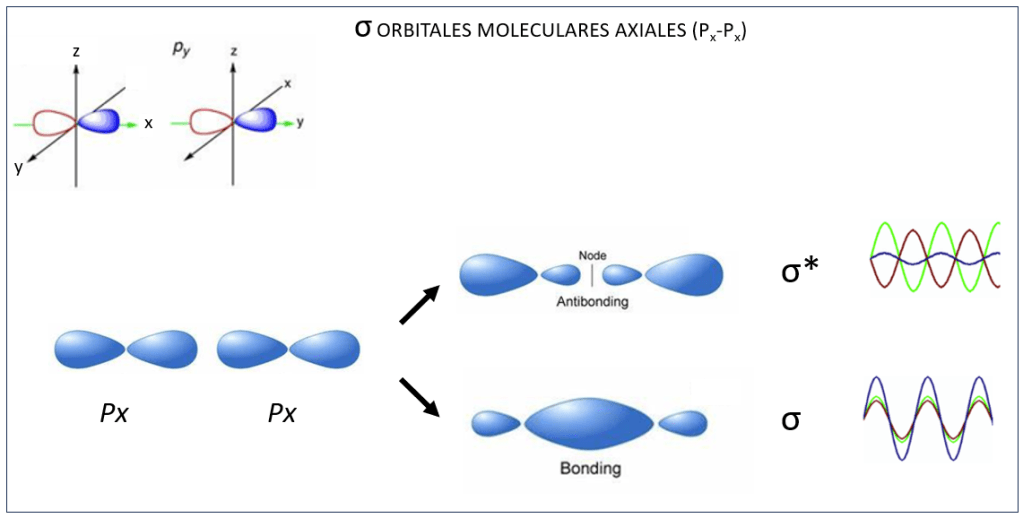
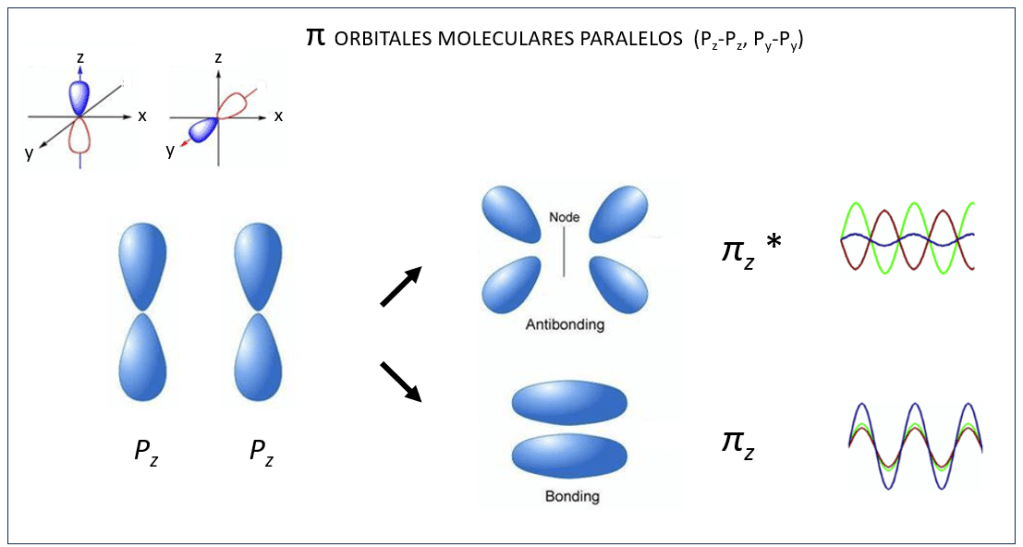




 , corresponden a los siguientes eigenspinors:
, corresponden a los siguientes eigenspinors: